La historia de la secuenciación unicelular.
La tecnología de secuenciación unicelular (SCS) se refiere a la secuenciación de la información genética transportada por las células a nivel de una sola célula, con el objetivo de obtener secuencias de genes, transcripciones, proteínas e información epigenética de un determinado tipo de célula y realizar el análisis integrado. Se ha utilizado ampliamente en la identificación de nuevas especies, la detección de patógenos, la evolución de patógenos, la biología del desarrollo, la neurociencia, la investigación de la heterogeneidad de los tumores y las células tumorales circulantes [1-2]. En 2013, la tecnología de secuenciación unicelular fue nombrada Tecnología del Año por Métodos de la naturaleza. En el mismo año, Ciencia clasificó la secuencia unicelular en la parte superior de las seis áreas más notables del año.
Desde la publicación del primer artículo de investigación del transcriptoma unicelular en 2009, la secuenciación unicelular ha sido objeto de más de diez años de desarrollo. Con la mejora continua de una variedad de tecnologías y la aparición de nuevas tecnologías, por ejemplo, la introducción de microfluidos, métodos de captura aleatoria y códigos de barras in situ ha promovido la reducción de costos de reactivos y consumibles, y la escala de secuenciación ha aumentado. también aumentó de ~100 células a cientos de miles o incluso millones de células.
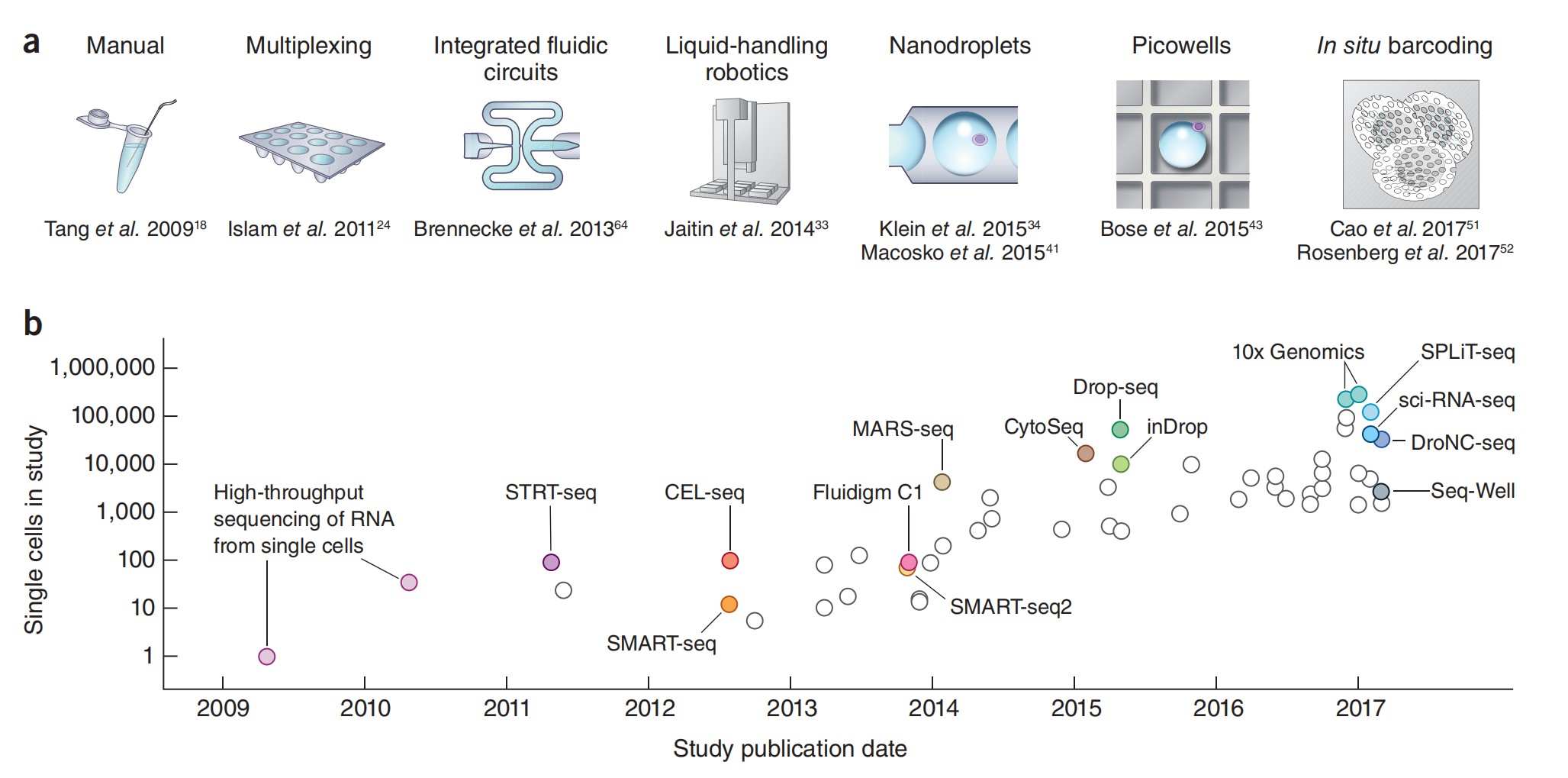
Figura 1: Escalado de experimentos de scRNA-seq.。(a) Tecnologías clave que han permitido saltos en la escala experimental; (b) Números de células informados en publicaciones representativas por fecha de publicación[3].
¿Cómo lograr la secuenciación a nivel celular?
Hay entre 40 y 60 billones de células en el cuerpo humano y el diámetro celular promedio está entre 5 y 200 micrones. Para secuenciar una sola célula, la primera estrategia que me viene a la mente es aislar la célula, construir una biblioteca de secuenciación de forma independiente y finalmente realizar la secuenciación. Existe una variedad de métodos que se pueden utilizar para manipular/separar y analizar con precisión células individuales, incluida la citometría de flujo, sistemas de microfluidos y varios métodos para separar y detectar células individuales en sistemas de micromódulos.
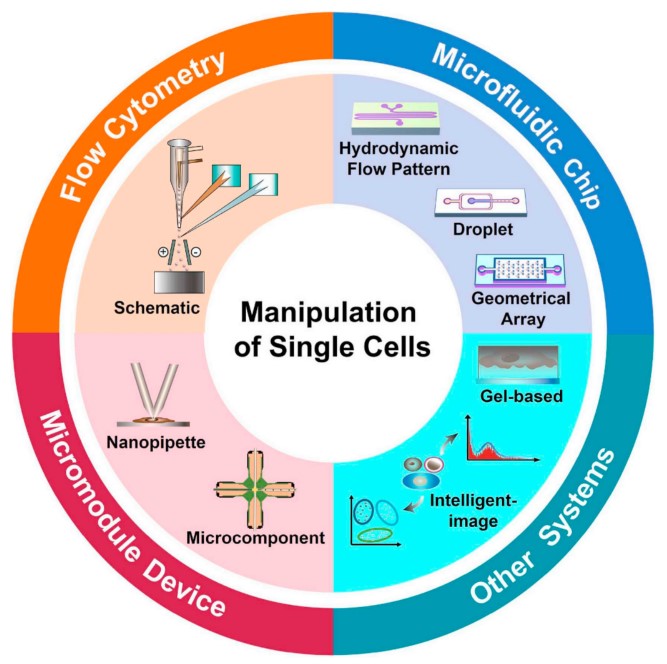
Figura 2: Clasificación de diversas estrategias para el aislamiento y manipulación de células individuales [4].
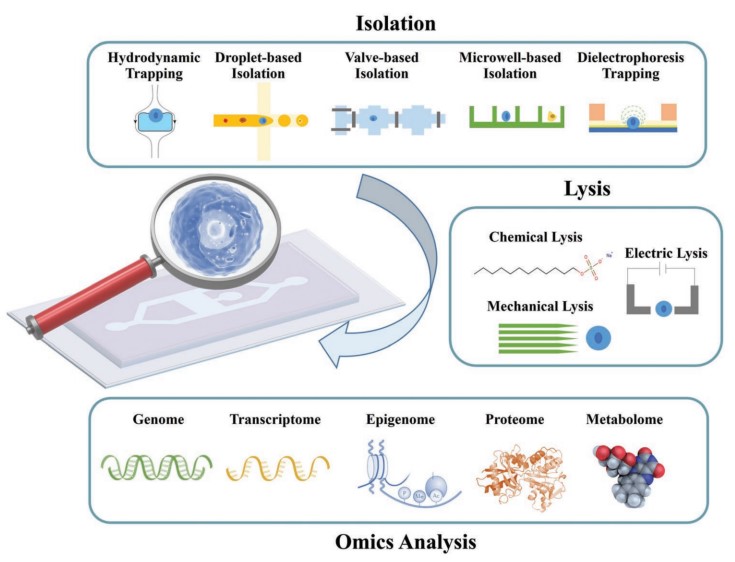
Figura 3: Ilustración esquemática del análisis ómico unicelular de microfluidos [5].
Sin embargo, esta estrategia ahorra muestras de células, pero el alto costo limita el aumento del rendimiento y su amplia aplicación. En los últimos años, las plataformas comerciales de secuenciación unicelular, como BD Rhapsody y 10X Genomics, han introducido la identificación unicelular basada en códigos de barras. Agregue un código de barras de secuencia de ADN único a cada célula y se podrá considerar que las moléculas de ácido nucleico que llevan la misma secuencia de ADN provienen de la misma célula. El uso de esta estrategia simplifica enormemente el proceso operativo de construcción de la biblioteca de secuenciación y se puede obtener la información de cientos de miles de células en la misma biblioteca.
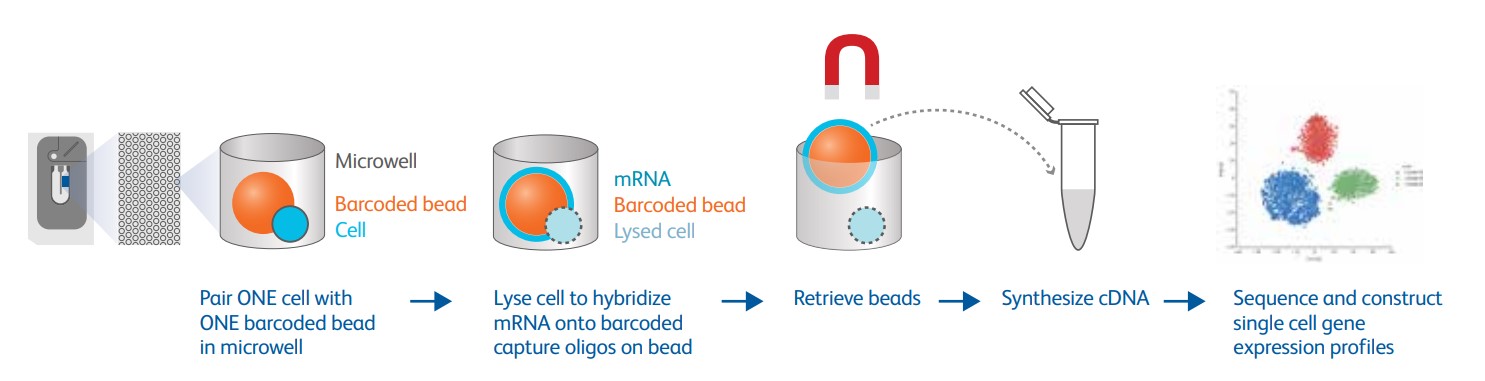
Figura 4: Flujo de trabajo de BD Rhapsody: un micropocillo = una célula = una perla magnética = un RNA-Seq (del sitio web oficial de BD)
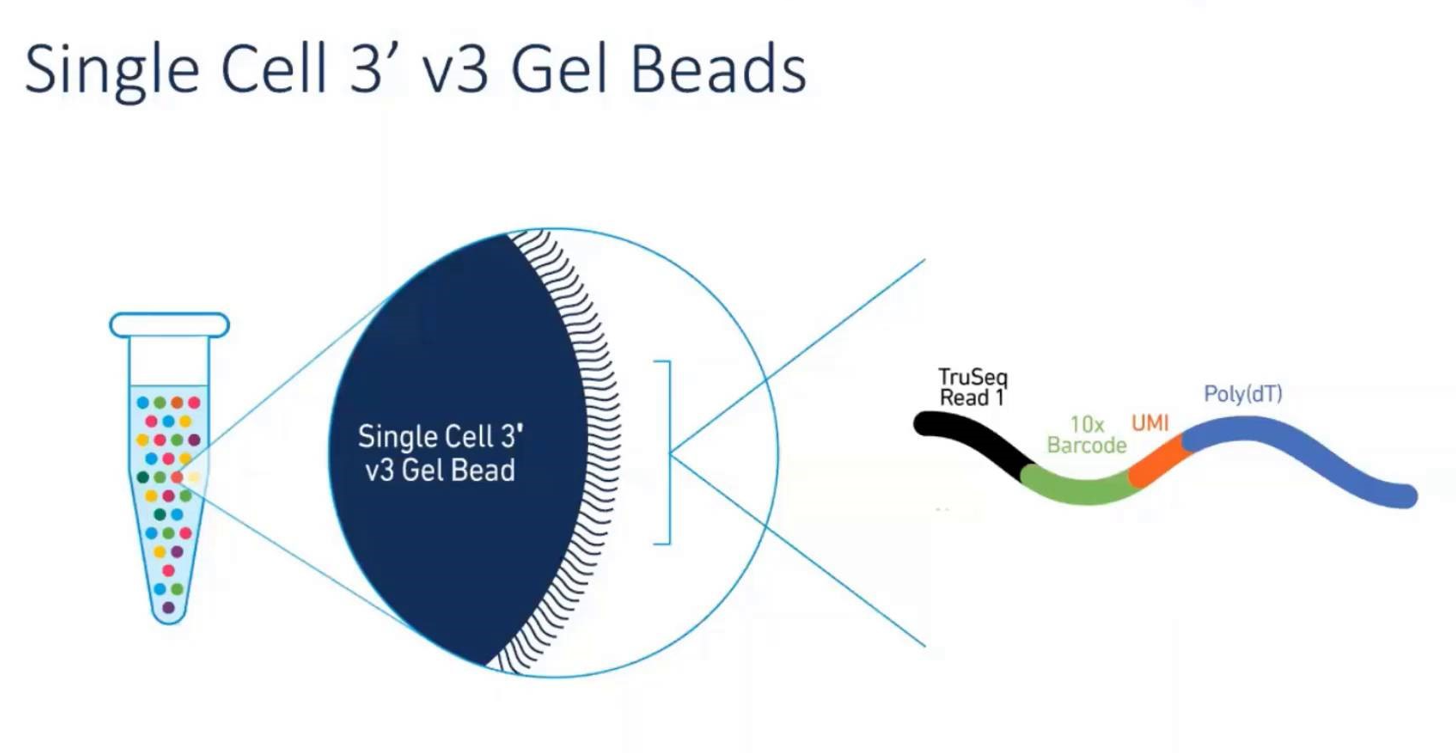
Figura 5: El flujo de trabajo de 10X Genomics: una gota de aceite = una célula = una perla de gel = una secuencia de ARN (del sitio web oficial de 10X Genomics)

